(no subject)
Nov. 12th, 2018 06:37 amЭто ответ sciuro про почему я расстраиваюсь, что в "науке" ИБД.
Может быть, дело в конкретной теме. У меня общее впечатление, что цитометрия и иммунология состоят из в основном дельных проектов. Еще у меня такое же ощущение про КРИСПР или другие, связанные с изготовлением плазмидов, например, темы. GFP-related stuff пока что тоже вроде сравнительно ок. В секвенировании _геномов_ все сравнительно ок. А вот в РНК-сек все очень плохо. И в куПЦР все очень, очень плохо. Про микроэррэи я вообще молчу, мне даже туда лезть не хочется, так там всё плохо.
Я могу попробовать объяснить. Я же тут последние три кода в Quantitative Biology Center и занимаюсь статистикой и квантификацией. И это очень, очень плохо. РНК-сек - это (во всяком случае пока) такая фигня, что уши сворачиваются в трубочку от ужаса, ЧТО люди выдают за реальные данные. Я для примера приведу две статьи, которые очень хорошо характеризуют состояние дел в этой области.
Вот одна статья. Делала ее никому неизвестная бедная лаба из области агрономии (люди с растениями - это вообще несчастные люди, у них как правило сильно мало денег, и к ним мало кто идет). Статья ПРЕКРАСНАЯ, просто золото. Написана максимально простым, логичным языком, видно, что у людей огромный опыт, что они не только специалисты, не только могут разобраться в данных, но и могут при этом внятно объяснить, как они это сделали, какие недостатки, как их надо воспринимать, и что можно сделать для решения проблем. Однако их прекрасная статья хрен знает где в каком жопо-журнале висит.
https://onlinelibrary.wiley.com/doi/epdf/10.1111/tpj.13014
Кратко: эти люди взяли и симулировали идеальный датасет, который получился бы при РНК-секе, если бы РНК-сек был сделан в идеальных условиях. Ну то есть "чистые данные", что никогда не бывает в реальных условиях. Далее, они взяли этот идеальный датасет, и прогнали его на доступных софтах для анализа РНК-сек данных. Как если бы они прогоняли реальный, грязный датасет. Так вот. ДАЖЕ ПРИ ИДЕАЛЬНОМ датасете, у них более 25% генов(!!!) "поехали" и дали кривой количественный результат, отклоняющийся на более чем 20% от реальных цифр (реальные цифры были известны, т.к. датасет был симулирован сознательно, и цифры были заложены создателями).
Четверть бля! Четверть всех генов поплыли! На ИДЕАЛЬНОМ датасете! А представьте что там в реале получается, когда люди приносят хрен знает какую грязную РНК, которая потом еще и напэцээрена сверху, потом из нее хрен знает как сделала библиотека хрен знает каким криворуким студентом...
Я щас покажу картинку, сколько критических ступенек в РНК-секе, на которых [количественный] результат может "поплыть":
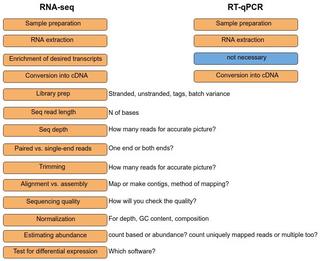
Слева РНК-сек ступени, на которых может поплыть. Из 14 ступеней только 13 и 14 - это анализ софтом. Все остальное может и будет вносить шум в количественное измерение. О каком в жопу точном измерении экспрессии можно говорить, простите меня пожалуйста? И это я еще НЕ ВКЛЮЧИЛА пэцээрение туда!!!! Которое при single-cell RNA-seq обязательно!!!
Важное уточнение: я не говорю о качестенном прочтении транскриптома, про качественное у меня ощущение, что там есть основания, т.к. секвенирование само по себе работает сравнительно ок, если что-то прочиталось, значит оно там как минимум было. Я говорю про количественное измерение экспрессии генов.
И спасибо большое агрономам с их статьей, с симулированным датасетом, который четко показал, насколько реально можно доверять анализу софтами... и это еще если учесть, что этих софтов понаплодилось - лопатой ешь... и все как один лучше другого, если верить их создателям((((
А теперь смотрите какая говностатья выложена в Нейчер (тут слово "говностатья", к сожалению, употреблено в прямом смысле, меня просто выворачивает на нее смотреть, настолько она страшно, некачественно написана, будто ее курица писала левой лапой):
https://www.ncbi.nlm.nih.gov/pubmed/19015660
Там ТАКИЕ дифирамбы РНК-секу, что у меня четкое ощущение, чтопост проплачен статья проплачена. В буквальном смысле проплачена, компаниями, выполняющими этот самый РНК-сек. Ну страшно читать, ей-богу, особенно после первой агростатьи. Просто страшно.
А она в Нейчер. И на нее ссылаются. И ее читают зеленые студенты. Ну мрак блин.
Может быть, дело в конкретной теме. У меня общее впечатление, что цитометрия и иммунология состоят из в основном дельных проектов. Еще у меня такое же ощущение про КРИСПР или другие, связанные с изготовлением плазмидов, например, темы. GFP-related stuff пока что тоже вроде сравнительно ок. В секвенировании _геномов_ все сравнительно ок. А вот в РНК-сек все очень плохо. И в куПЦР все очень, очень плохо. Про микроэррэи я вообще молчу, мне даже туда лезть не хочется, так там всё плохо.
Я могу попробовать объяснить. Я же тут последние три кода в Quantitative Biology Center и занимаюсь статистикой и квантификацией. И это очень, очень плохо. РНК-сек - это (во всяком случае пока) такая фигня, что уши сворачиваются в трубочку от ужаса, ЧТО люди выдают за реальные данные. Я для примера приведу две статьи, которые очень хорошо характеризуют состояние дел в этой области.
Вот одна статья. Делала ее никому неизвестная бедная лаба из области агрономии (люди с растениями - это вообще несчастные люди, у них как правило сильно мало денег, и к ним мало кто идет). Статья ПРЕКРАСНАЯ, просто золото. Написана максимально простым, логичным языком, видно, что у людей огромный опыт, что они не только специалисты, не только могут разобраться в данных, но и могут при этом внятно объяснить, как они это сделали, какие недостатки, как их надо воспринимать, и что можно сделать для решения проблем. Однако их прекрасная статья хрен знает где в каком жопо-журнале висит.
https://onlinelibrary.wiley.com/doi/epdf/10.1111/tpj.13014
Кратко: эти люди взяли и симулировали идеальный датасет, который получился бы при РНК-секе, если бы РНК-сек был сделан в идеальных условиях. Ну то есть "чистые данные", что никогда не бывает в реальных условиях. Далее, они взяли этот идеальный датасет, и прогнали его на доступных софтах для анализа РНК-сек данных. Как если бы они прогоняли реальный, грязный датасет. Так вот. ДАЖЕ ПРИ ИДЕАЛЬНОМ датасете, у них более 25% генов(!!!) "поехали" и дали кривой количественный результат, отклоняющийся на более чем 20% от реальных цифр (реальные цифры были известны, т.к. датасет был симулирован сознательно, и цифры были заложены создателями).
Четверть бля! Четверть всех генов поплыли! На ИДЕАЛЬНОМ датасете! А представьте что там в реале получается, когда люди приносят хрен знает какую грязную РНК, которая потом еще и напэцээрена сверху, потом из нее хрен знает как сделала библиотека хрен знает каким криворуким студентом...
Я щас покажу картинку, сколько критических ступенек в РНК-секе, на которых [количественный] результат может "поплыть":

Слева РНК-сек ступени, на которых может поплыть. Из 14 ступеней только 13 и 14 - это анализ софтом. Все остальное может и будет вносить шум в количественное измерение. О каком в жопу точном измерении экспрессии можно говорить, простите меня пожалуйста? И это я еще НЕ ВКЛЮЧИЛА пэцээрение туда!!!! Которое при single-cell RNA-seq обязательно!!!
Важное уточнение: я не говорю о качестенном прочтении транскриптома, про качественное у меня ощущение, что там есть основания, т.к. секвенирование само по себе работает сравнительно ок, если что-то прочиталось, значит оно там как минимум было. Я говорю про количественное измерение экспрессии генов.
И спасибо большое агрономам с их статьей, с симулированным датасетом, который четко показал, насколько реально можно доверять анализу софтами... и это еще если учесть, что этих софтов понаплодилось - лопатой ешь... и все как один лучше другого, если верить их создателям((((
А теперь смотрите какая говностатья выложена в Нейчер (тут слово "говностатья", к сожалению, употреблено в прямом смысле, меня просто выворачивает на нее смотреть, настолько она страшно, некачественно написана, будто ее курица писала левой лапой):
https://www.ncbi.nlm.nih.gov/pubmed/19015660
Там ТАКИЕ дифирамбы РНК-секу, что у меня четкое ощущение, что
А она в Нейчер. И на нее ссылаются. И ее читают зеленые студенты. Ну мрак блин.
no subject
Date: 2018-11-11 11:36 pm (UTC)Ðа, ÑÑÐ¾Ñ Ð¿Ð¾ÑÑ Ð¿Ð¾-английÑки назÑваеÑÑÑ preaching to the choir, Ñ Ð½Ð° наÑей пÑоÑлой болÑÑой конÑеÑенÑии пÑоводила Ñ Ñолпой дÑÑÐ³Ð¸Ñ Ð²ÐµÑелÑÑ ÑанаÑиков
ÐºÐ¾Ð¼Ñ Ð½ÐµÑего ÑеÑÑÑÑдиÑкÑÑÑионнÑÑ ÑекÑÐ¸Ñ Ð¿Ñо Ñо, ÑколÑко пÑоблем Ñ scRNA-seq :)ÐÑлиÑÐ½Ð°Ñ ÑÑаÑÑÑ Ð°Ð³Ñономов, одно Ð¼Ð¾Ð³Ñ ÑказаÑÑ - она 2015 года, по меÑкам ÑÑой облаÑÑи - пÑоÑлÑй век. Ð ÑÑаÑÑÑ Ð² нейÑÑÑ ÐµÑе дÑевнее. Рда, ÑÑаÑÑи в Nature Reviews Anything вÑе пиÑÑÑÑÑ Ð¿Ð¾ пÑиглаÑениÑ, не ÑамоÑеком. ÐÑо не компании, но да, ÑÑÑ ÑÑаÑÑÑ Ð·Ð°ÐºÐ°Ð·Ð°Ð»Ð¸ ÑÑим авÑоÑам, поÑÐ¾Ð¼Ñ ÑÑо ÑедакÑÐ¸Ñ Ñ Ð¾Ñела заÑикÑиÑоваÑÑ Ð¼Ð½ÐµÐ½Ð¸Ðµ конкÑеÑно ÑÑой гÑÑÐ¿Ð¿Ñ Ð¸Ð· ÐелÑ.
no subject
Date: 2018-11-12 05:30 am (UTC)ÐÐ¾Ð¶ÐµÑ ÑÑÑдноÑÑи пеÑевода.
no subject
Date: 2018-11-12 06:25 am (UTC)no subject
Date: 2018-11-12 09:46 am (UTC)Ðогда ÑазбиваÑÑ ÑÑнкÑиониÑование клеÑки на оÑделÑнÑе более-менее обоÑобленнÑе пÑоÑеÑÑÑ, когда вÑделÑÑÑ ÐºÐ°ÐºÐ¸Ðµ-Ñо молекÑлÑÑнÑе комплекÑÑ, оÑвеÑÑÑвеннÑе за Ñо, и за ÑÑо.
ЧеÑÑно ÑказаÑÑ, менÑ, как Ñ Ð¸Ð¼Ð¸ÐºÐ°, ÑÑо пÑÐ¸Ð²Ð¾Ð´Ð¸Ñ Ð² оÑÑÑение где-Ñо пÑомежÑÑоÑное Ð¼ÐµÐ¶Ð´Ñ Ð±Ð»Ð°Ð³Ð¾Ð³Ð¾Ð²ÐµÐ½Ð¸ÐµÐ¼ и оÑÑиÑанием. СкладÑваеÑÑÑ Ð²Ð¿ÐµÑаÑление, ÑÑо либо нам оÑÐµÐ½Ñ Ð¿Ð¾Ð²ÐµÐ·Ð»Ð¾ Ñ ÑÑÑÑойÑÑвом клеÑки, либо вÑе наÑе понимание ÑÑо smoke and mirrors.
no subject
Date: 2018-11-12 10:08 am (UTC)no subject
Date: 2018-11-12 10:13 am (UTC)Ð Ð´Ð»Ñ Ð½Ð°ÑÑоÑÑей наÑки (как и Ð´Ð»Ñ Ð²Ñего оÑÑалÑного наÑÑоÑÑего) еÑÑÑ Ð¶Ð¾Ð¿Ð¾-жÑÑналÑ. Самое главное доÑÑижение в биологии поÑледней ÑоÑни Ð»ÐµÑ Ð²Ð¾Ð¾Ð±Ñе виÑÐ¸Ñ Ð½Ð° одной ÑÑÑемной ÑÑÑаниÑе в инÑеÑнеÑе, пÑоÑиÑанное паÑой ÑÑÑÑÑ ÑлÑÑайнÑÑ Ð»Ñдей.
no subject
Date: 2018-11-12 01:24 pm (UTC)no subject
Date: 2018-11-12 02:16 pm (UTC)no subject
Date: 2018-11-12 03:17 pm (UTC)no subject
Date: 2018-11-12 03:21 pm (UTC)no subject
Date: 2018-11-12 02:33 pm (UTC)no subject
Date: 2018-11-12 02:34 pm (UTC)no subject
Date: 2018-11-15 08:09 am (UTC)no subject
Date: 2018-11-15 11:42 am (UTC)no subject
Date: 2018-11-13 07:56 pm (UTC)ÐаÑод Ñ Ð¾ÑÐµÑ Ð·Ð½Ð°ÑÑ. Ðегоже деÑжаÑÑ Ð·Ð½Ð°Ð½Ð¸Ñ Ð² Ñайне.
no subject
Date: 2018-11-13 09:44 pm (UTC)no subject
Date: 2018-11-14 03:44 am (UTC)поÑÑÐ¾Ð¼Ñ Ð¸ ÑиÑаеÑÑÑ "паÑой ÑÑÑÑÑ ÑлÑÑайнÑÑ Ð»Ñдей". Ð ÑÐµÐ¼Ñ ÑÐ°ÐºÐ°Ñ ÑекÑеÑноÑÑÑ?
no subject
Date: 2018-11-14 08:39 am (UTC)РпÑÑаÑÑÑÑ Ð±Ð¾ÑоÑÑÑÑ Ñ Ñем, ÑÑо "заинÑеÑеÑованнÑе" лÑди Ñже не гоÑÐ¾Ð²Ñ Ð»Ð¸Ñние паÑÑ Ñлов в поиÑковик забиÑÑ, ÑÑо какое-Ñо полное неÑважение к Ñебе.
no subject
Date: 2018-11-12 08:47 pm (UTC)У нейÑÑÑов/ÑÑаенÑов ÑовеÑÑенно ÑаздÑÑÑй IF, но Ð³Ð¾Ð´Ð½Ð°Ñ Ð½Ð°Ñка, как Ñаз ÑакаÑ, как вам нÑавиÑÑÑ - без ÑпеÑки, без ÑаздÑÑÑÑ ÑезÑлÑÑаÑов - как Ñаз пÑбликÑеÑÑÑ Ð² коÑидоÑе IF 3-12. Ðногда и в нейÑÑÑ Ñоже :)
no subject
Date: 2018-11-13 03:47 pm (UTC)ÐлÑÑ, вÑем же Ñже давно вÑе Ñавно, где напеÑаÑано? ÐÑ Ñо еÑÑÑ Ð´ÐµÐ»Ð°ÐµÑÑ ÐºÐ¾Ð½ÑÑÐ¾Ð»Ñ Ð½Ð° дÑиÑÑ Ð¸Ð· жÑÑналов-Ñ Ð¸Ñников, а Ñам вÑе Ñавно ÑиÑаеÑÑ Ð°Ð±ÑÑÑÐ°ÐºÑ Ð¸ Ð¼Ð¾Ð¶ÐµÑ Ð¿Ð¾Ñом ÑÐ°Ð¼Ñ ÑÑаÑÑÑ ÐµÑли ÑелеванÑно.
То еÑÑÑ Ð²Ð°Ð¶Ð½Ð¾ какой жÑÑнал, конеÑно - из ÑообÑажений за наÑколÑко зловÑеднÑм пÑйволлом оно лежиÑ, Ð´Ð»Ñ ÑÐµÑ ÐºÑо пÑо либген не Ð·Ð½Ð°ÐµÑ (и Ð´Ð»Ñ ÑÑаÑÑÑ Ð¸ ÑкзоÑиÑеÑÐºÐ¸Ñ ÑÑаÑей).
no subject
Date: 2018-11-20 12:39 am (UTC)к 2020 Ð³Ð¾Ð´Ñ ÐСРнаÑÑнÑе пÑбликаÑии, издание коÑоÑÑÑ ÑпонÑиÑÑеÑÑÑ Ð³Ð¾ÑÑдаÑÑÑвеннÑми Ñондами в ÐС (90%+ вÑÐµÑ ÑабоÑ) Ð¿Ð¾Ð´Ð»ÐµÐ¶Ð¸Ñ ÑазмеÑÐµÐ½Ð¸Ñ Ð½Ð° плаÑÑоÑÐ¼Ð°Ñ Ð¸/или жÑÑÐ½Ð°Ð»Ð°Ñ Ñ Ð¾ÑкÑÑÑÑм доÑÑÑпом.
no subject
Date: 2018-12-20 08:20 pm (UTC)So. we fix the most interesting and we look: what is THE MOST interesting
It's a logic
But, the world isn't normal
So, you see
no subject
Date: 2018-12-20 09:13 pm (UTC)Money is a fun.
There is no way for any reasonable person to have less money then you need to survive.
No way.
The problem is not to have TOO HUGE amount of money.
Scientists ARE reasonable persons.
Nobody expect them in abscence of money.
So, they should pay for fixation, because we don't want being fixed any nonsense like abstraction.
And...
Nobody expected this wars for money. War is also a fun, basically. But...too much deaths.