(no subject)
Nov. 12th, 2018 06:37 amЭто ответ sciuro про почему я расстраиваюсь, что в "науке" ИБД.
Может быть, дело в конкретной теме. У меня общее впечатление, что цитометрия и иммунология состоят из в основном дельных проектов. Еще у меня такое же ощущение про КРИСПР или другие, связанные с изготовлением плазмидов, например, темы. GFP-related stuff пока что тоже вроде сравнительно ок. В секвенировании _геномов_ все сравнительно ок. А вот в РНК-сек все очень плохо. И в куПЦР все очень, очень плохо. Про микроэррэи я вообще молчу, мне даже туда лезть не хочется, так там всё плохо.
Я могу попробовать объяснить. Я же тут последние три кода в Quantitative Biology Center и занимаюсь статистикой и квантификацией. И это очень, очень плохо. РНК-сек - это (во всяком случае пока) такая фигня, что уши сворачиваются в трубочку от ужаса, ЧТО люди выдают за реальные данные. Я для примера приведу две статьи, которые очень хорошо характеризуют состояние дел в этой области.
Вот одна статья. Делала ее никому неизвестная бедная лаба из области агрономии (люди с растениями - это вообще несчастные люди, у них как правило сильно мало денег, и к ним мало кто идет). Статья ПРЕКРАСНАЯ, просто золото. Написана максимально простым, логичным языком, видно, что у людей огромный опыт, что они не только специалисты, не только могут разобраться в данных, но и могут при этом внятно объяснить, как они это сделали, какие недостатки, как их надо воспринимать, и что можно сделать для решения проблем. Однако их прекрасная статья хрен знает где в каком жопо-журнале висит.
https://onlinelibrary.wiley.com/doi/epdf/10.1111/tpj.13014
Кратко: эти люди взяли и симулировали идеальный датасет, который получился бы при РНК-секе, если бы РНК-сек был сделан в идеальных условиях. Ну то есть "чистые данные", что никогда не бывает в реальных условиях. Далее, они взяли этот идеальный датасет, и прогнали его на доступных софтах для анализа РНК-сек данных. Как если бы они прогоняли реальный, грязный датасет. Так вот. ДАЖЕ ПРИ ИДЕАЛЬНОМ датасете, у них более 25% генов(!!!) "поехали" и дали кривой количественный результат, отклоняющийся на более чем 20% от реальных цифр (реальные цифры были известны, т.к. датасет был симулирован сознательно, и цифры были заложены создателями).
Четверть бля! Четверть всех генов поплыли! На ИДЕАЛЬНОМ датасете! А представьте что там в реале получается, когда люди приносят хрен знает какую грязную РНК, которая потом еще и напэцээрена сверху, потом из нее хрен знает как сделала библиотека хрен знает каким криворуким студентом...
Я щас покажу картинку, сколько критических ступенек в РНК-секе, на которых [количественный] результат может "поплыть":
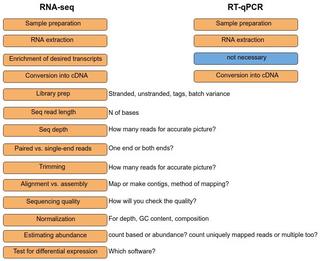
Слева РНК-сек ступени, на которых может поплыть. Из 14 ступеней только 13 и 14 - это анализ софтом. Все остальное может и будет вносить шум в количественное измерение. О каком в жопу точном измерении экспрессии можно говорить, простите меня пожалуйста? И это я еще НЕ ВКЛЮЧИЛА пэцээрение туда!!!! Которое при single-cell RNA-seq обязательно!!!
Важное уточнение: я не говорю о качестенном прочтении транскриптома, про качественное у меня ощущение, что там есть основания, т.к. секвенирование само по себе работает сравнительно ок, если что-то прочиталось, значит оно там как минимум было. Я говорю про количественное измерение экспрессии генов.
И спасибо большое агрономам с их статьей, с симулированным датасетом, который четко показал, насколько реально можно доверять анализу софтами... и это еще если учесть, что этих софтов понаплодилось - лопатой ешь... и все как один лучше другого, если верить их создателям((((
А теперь смотрите какая говностатья выложена в Нейчер (тут слово "говностатья", к сожалению, употреблено в прямом смысле, меня просто выворачивает на нее смотреть, настолько она страшно, некачественно написана, будто ее курица писала левой лапой):
https://www.ncbi.nlm.nih.gov/pubmed/19015660
Там ТАКИЕ дифирамбы РНК-секу, что у меня четкое ощущение, чтопост проплачен статья проплачена. В буквальном смысле проплачена, компаниями, выполняющими этот самый РНК-сек. Ну страшно читать, ей-богу, особенно после первой агростатьи. Просто страшно.
А она в Нейчер. И на нее ссылаются. И ее читают зеленые студенты. Ну мрак блин.
Может быть, дело в конкретной теме. У меня общее впечатление, что цитометрия и иммунология состоят из в основном дельных проектов. Еще у меня такое же ощущение про КРИСПР или другие, связанные с изготовлением плазмидов, например, темы. GFP-related stuff пока что тоже вроде сравнительно ок. В секвенировании _геномов_ все сравнительно ок. А вот в РНК-сек все очень плохо. И в куПЦР все очень, очень плохо. Про микроэррэи я вообще молчу, мне даже туда лезть не хочется, так там всё плохо.
Я могу попробовать объяснить. Я же тут последние три кода в Quantitative Biology Center и занимаюсь статистикой и квантификацией. И это очень, очень плохо. РНК-сек - это (во всяком случае пока) такая фигня, что уши сворачиваются в трубочку от ужаса, ЧТО люди выдают за реальные данные. Я для примера приведу две статьи, которые очень хорошо характеризуют состояние дел в этой области.
Вот одна статья. Делала ее никому неизвестная бедная лаба из области агрономии (люди с растениями - это вообще несчастные люди, у них как правило сильно мало денег, и к ним мало кто идет). Статья ПРЕКРАСНАЯ, просто золото. Написана максимально простым, логичным языком, видно, что у людей огромный опыт, что они не только специалисты, не только могут разобраться в данных, но и могут при этом внятно объяснить, как они это сделали, какие недостатки, как их надо воспринимать, и что можно сделать для решения проблем. Однако их прекрасная статья хрен знает где в каком жопо-журнале висит.
https://onlinelibrary.wiley.com/doi/epdf/10.1111/tpj.13014
Кратко: эти люди взяли и симулировали идеальный датасет, который получился бы при РНК-секе, если бы РНК-сек был сделан в идеальных условиях. Ну то есть "чистые данные", что никогда не бывает в реальных условиях. Далее, они взяли этот идеальный датасет, и прогнали его на доступных софтах для анализа РНК-сек данных. Как если бы они прогоняли реальный, грязный датасет. Так вот. ДАЖЕ ПРИ ИДЕАЛЬНОМ датасете, у них более 25% генов(!!!) "поехали" и дали кривой количественный результат, отклоняющийся на более чем 20% от реальных цифр (реальные цифры были известны, т.к. датасет был симулирован сознательно, и цифры были заложены создателями).
Четверть бля! Четверть всех генов поплыли! На ИДЕАЛЬНОМ датасете! А представьте что там в реале получается, когда люди приносят хрен знает какую грязную РНК, которая потом еще и напэцээрена сверху, потом из нее хрен знает как сделала библиотека хрен знает каким криворуким студентом...
Я щас покажу картинку, сколько критических ступенек в РНК-секе, на которых [количественный] результат может "поплыть":

Слева РНК-сек ступени, на которых может поплыть. Из 14 ступеней только 13 и 14 - это анализ софтом. Все остальное может и будет вносить шум в количественное измерение. О каком в жопу точном измерении экспрессии можно говорить, простите меня пожалуйста? И это я еще НЕ ВКЛЮЧИЛА пэцээрение туда!!!! Которое при single-cell RNA-seq обязательно!!!
Важное уточнение: я не говорю о качестенном прочтении транскриптома, про качественное у меня ощущение, что там есть основания, т.к. секвенирование само по себе работает сравнительно ок, если что-то прочиталось, значит оно там как минимум было. Я говорю про количественное измерение экспрессии генов.
И спасибо большое агрономам с их статьей, с симулированным датасетом, который четко показал, насколько реально можно доверять анализу софтами... и это еще если учесть, что этих софтов понаплодилось - лопатой ешь... и все как один лучше другого, если верить их создателям((((
А теперь смотрите какая говностатья выложена в Нейчер (тут слово "говностатья", к сожалению, употреблено в прямом смысле, меня просто выворачивает на нее смотреть, настолько она страшно, некачественно написана, будто ее курица писала левой лапой):
https://www.ncbi.nlm.nih.gov/pubmed/19015660
Там ТАКИЕ дифирамбы РНК-секу, что у меня четкое ощущение, что
А она в Нейчер. И на нее ссылаются. И ее читают зеленые студенты. Ну мрак блин.